การวิเคราะห์ข้อมูลที่มีการวางแแผนการทดลองแบบ CRD ด้วย R ดัพเดท ปี 2026
08-09-2023
สำหรับการทดลงแบบ CRD ในยุคปัจจุบันนี้ สนับสนุนให้การรายงานควรที่จะแสดง “ค่าประมาณ + 95% CI” และ ขนาดเอฟเฟกต์ (η²/ω²) มากกว่าดูจากค่า p-value อย่างเดียว
พยายามรวบรวม วิธีการวิเคราะห์ข้อมูลสถิติกับการทดลองที่ออกแบบ แบบ CRD ด้วย R code โดยพยายามให้โค้ดมีความทันสมัยและอยู่ในรูปแบบการโค้ด package ที่เป็นปัจจุบัน มชมากที่สุด
เรามาเริ่มกันโค้ดเลย
1. โหลด package ที่จำเป็น
# packages
pacman::p_load(
tidyverse, # data import and handling
conflicted, # handling function conflicts
emmeans,
multcomp,
multcompView, # adjusted mean comparisons
ggplot2,
effectsize, # effect size calculation
desplot, # for plotting experimental designs
rstatix,
ggdist,
performance,
see
) # plots
# conflicts between functions with the same name
conflict_prefer("filter", "dplyr")
conflict_prefer("select", "dplyr")
pacman::p_load เป็น package ที่ช่วยให้เราโหลด package ได้ง่ายขึ้น โดยไม่ต้องโหลดทีละตัว
tidyverse ช่วยจัดการข้อมูลได้ง่ายขึ้น conflicted ช่วยจัดการ function conflicts ซึ่ง เมื่อประกาศใช้ tidyverse แล้ว จะมี function ซ้ำกันหลายตัวกับ R base อยู่ เพื่อไม่ให้เกิดความขัดแย้งกันเองก็เลือกเลยว่าจะให้อะไรจาก package ไหน
emmeans ช่วยให้เราจัดการ adjusted mean comparisons ได้ง่ายขึ้น multcomp ช่วยให้เราจัดการ adjusted mean comparisons ได้ง่ายขึ้น multcompView ช่วยให้เราจัดการ adjusted mean comparisons ได้ง่ายขึ้น effectsize ช่วยให้เราจัดการ effect size calculation ได้ง่ายขึ้น
เพื่อตรวจสอบสมมุติฐานของ ANOVA
desplot ช่วยให้เราจัดการ for plotting experimental designs ได้ง่ายขึ้น
rstatix ช่วยให้เราจัดการ rstatix ได้ง่ายขึ้น
ggdist ช่วยให้เราจัดการ ggdist ได้ง่ายขึ้น
performance ช่วยให้เราจัดการ performance ได้ง่ายขึ้น
see ช่วยให้เราจัดการ see ได้ง่ายขึ้น
2. การนำเข้าข้อมูล
ตัวอย่างข้อมูลที่นำมาให้ใช้ เป็น ข้อมูลที่มาทำได้เป็นงานวิจัยทดสอบผลผลิตเมลอน ตีพิมพ์ Mead et al. (1993, p.52) โดยการทดลองมีพันธุ์เมลอน 4 พันธุ์ แต่ละพันธุ์ได้รับการทดสอบในแปลงทดลองจำนวน 6 แปลง ออกแบบการทดสอบแบบ สุ่มสมบูรณ์ (CRD) จาก “Example 4.3” หนังสือ “Quantitative Methods in Biosciences (3402-420)” by Prof. Dr. Hans-Peter Piepho
#| label : "Import data"
dataURL <- "https://raw.githubusercontent.com/SchmidtPaul/DSFAIR/master/data/Mead1993.csv"
dat <- read_csv(dataURL)
dat %>% head()
ผลลัพธ์ที่ได้
variety yield row col
<chr> <dbl> <dbl> <dbl>
1 v1 25.1 4 2
2 v1 17.2 1 6
3 v1 26.4 4 1
4 v1 16.1 1 4
และต่อไปเราเพื่อที่จะวิเคราะห์ ANOVA จะต้องแปลง variety ให้เป็น factor ก่อน
dat <- dat %>%
mutate(variety = as.factor(variety))
ได้
# A tibble: 24 × 4
variety yield row col
<fct> <dbl> <dbl> <dbl>
1 v1 25.1 4 2
2 v1 17.2 1 6
3 v1 26.4 4 1
4 v1 16.1 1 4
ขอดูกราฟก่อนว่า หน้าตาของข้อมูลเป็นอย่างไร
ggplot(data = dat, aes(y = yield, x = variety)) +
geom_point() + # scatter plot
ylim(0, NA) + # force y-axis to start at 0
theme_classic() # clearer plot format
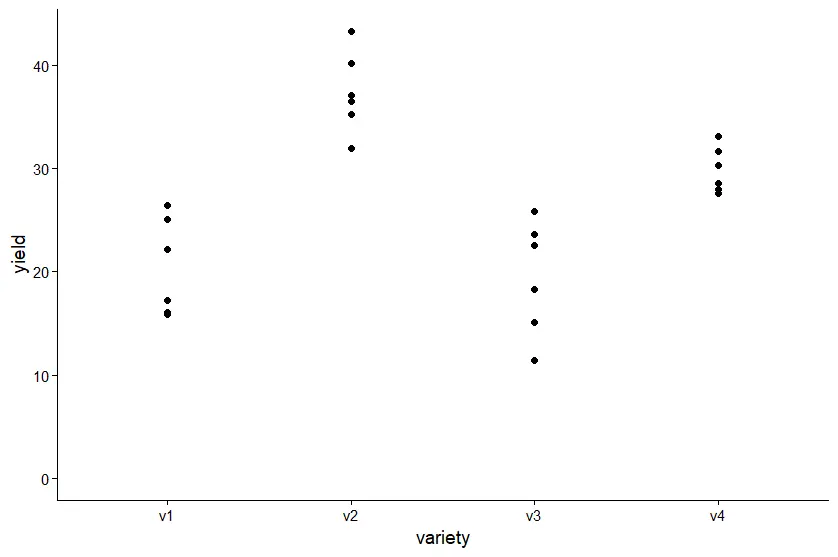
จากตรงนี้ก็พอจะเห็นอะไรบ้างว่าพันธุ์แต่ละพันธุ์นั้น มีแนวโน้มที่ให้ผลผลิตที่แตกต่างกัน
3. วิเคราะห์ ANOVA
อันนี้แหละ ที่เป้นประเด็น ในการเรื่องการวิเคราะห์ ANOVA Modern ANOVA = car::Anova(type = 3)
fit <- lm(yield ~ variety, data = dat)
car::Anova(fit, type = 3)
ถ้าเป็น code แบบ classic ก็ใช้ aov(yield ~ variety, data = dat) ซึ่ง ถ้าเป็น ปัจจุบันตอนนี้ เราจะนิยมและปลอดภัยสุด หรือ ANOVA type จาก Data Science for Agriculture in R
In most cases it’s probably best to conduct a
Type IIIANOVA, e.g. viacar::Anova(model, type = "III").
classic ANOVA
แบบ ANOVA จาก R base ก็ใช้ได้เช่นกัน นะ
#| eval : false
aov.model <- aov(yield ~ variety, data = dat)
summary(aov.model)
ถ้า ไม่ sig เราก็ จะรายงาน เท่านี้ แต่ถ้าไม่ ส่วนใหญ่ เราก็จะไปทดสอบต่อ อย่างที่เรา เรียกกัน postpoc
ตรวจ Asusumption
ใช้ performance library หรือ easystat library เพื่อ check สมมุติฐาน และคุณภาพของข้อมูล ที่นำไปวิเคราะห์ ANOVA
- Homogeneity check
performance::check_homogeneity(fit)
performance::check_homogeneity(fit) %>% plot()
- heteroscedasticit check
performance::check_heteroscedasticity(fit)
performance::check_heteroscedasticity(fit) %>% plot()
บอกว่าต้องเชคอันนี้ก่อน เพราะว่ามันจะมีผลต่อ การวิเคราะห์ ANOVA เพราะว่า ถ้าไม่ ก็ ควรใช้ Welch ANOVA แทน
- Normality check
performance::check_normality(fit)
performance::check_normality(fit) %>% plot()
หลายคนอยากดู outlier
check_outliers(fit) %>% plot()
ขนาดเอฟเฟกต์
อยากจะชวนให้ ดู effect ต่อ สำคัญอย่างไร - p-value บอกแค่ว่า “มีหลักฐานต่างกันไหม” ซึ่งไวต่อ ขนาดตัวอย่าง มาก แต่ ไม่บอกว่าผลที่ได้นั้นต่างกันมากขนาดไหน - ขนาดเอฟเฟกต์บอก “ผลที่ได้ใหญ่พอจะมีความหมายเชิงปฏิบัติไหม” และทำให้ เปรียบเทียบงาน/การทดลองต่างกันได้ บนมาตรฐานเดียว
effectsize::eta_squared(fit)
effectsize::omega_squared(fit)
ค่า omega square (variance explained): “ทรีตเมนต์อธิบายความแปรปรวนได้ ~73% (ω²=0.73) ผลต่างเห็นได้ชัด (รู้สึกมั่นใจว่าผลการทดลองที่ได้สังเกตเห็นผลต่างได้ชัดเจน)”
post-hoc test
แม้ปัจจุบัน นักสถิติหลายคน ไม่ค่อย สนับสนุนให้ ทำ CLD(compact letter dusplay) เพราะว่า … แล้ว แต่ในสาขาเกษตรหรือชีววิทยา ก็ยังคงนิยมเพระาว่า ง่ายในการอธิบายผล ดังนั้น เราจะยังใช้
ประมาณค่าเฉลี่ยเปรียบเทียบ โดยการใช้ตัวอักษร
emm <- emmeans(fit, ~variety)
contrast(emm, "pairwise", adjust = "tukey")
ตารางข้างบ้นนี้ เปรียบเทียบทีละคู่ ๆ
เปรียบเทียบโดยการใช้ตัวอักษร CLD
mean_comparisons <- fit %>%
emmeans(specs = "variety") %>%
cld(adjust = "tukey", Letters = letters)
mean_comparisons
กราฟเปรียบเทียบตัวแปร
ggplot(mean_comparisons, aes(variety, emmean)) +
ggdist::geom_pointinterval(
aes(ymin = lower.CL, ymax = upper.CL)
) +
labs(
x = "Treatment",
y = "Estimated mean (95% CI)",
title = "CRD: EMMs + 95% CI"
) +
theme_minimal(base_size = 13)
my_caption <- "Black dots = raw data (jittered). Red dots/bars = adjusted means with 95% CIs per variety. Letters share = not different (Tukey)."
ggplot() +
aes(x = variety) +
# จุดดิบ: jitter ป้องกันทับกัน + alpha ให้โปร่ง
geom_point(
data = dat,
aes(y = yield),
position = position_jitter(width = 0.08, height = 0),
alpha = 0.6,
size = 1.9
) +
# ค่ากลางและช่วงเชื่อมั่น (ใช้ pointrange แทน point + errorbar แยก)
geom_pointrange(
data = mean_comparisons,
aes(y = emmean, ymin = lower.CL, ymax = upper.CL),
color = "red",
fatten = 1.1,
size = 0.6,
position = position_nudge(x = 0.12)
) +
# ตัวอักษร CLD: ขยับไปขวาอีกหน่อย และยกขึ้นเล็กน้อย
geom_text(
data = mean_comparisons,
aes(y = emmean, label = .group),
color = "red",
position = position_nudge(x = 0.24),
hjust = 0,
vjust = -0.3,
fontface = "bold",
size = 3.6
) +
# แกนและสเกล
scale_x_discrete(name = "Variety", expand = expansion(mult = c(0.02, 0.22))) +
scale_y_continuous(
name = "Yield",
limits = c(0, NA),
expand = expansion(mult = c(0, 0.08))
) +
# ธีมสะอาด + ระยะขอบเผื่อข้อความ
theme_classic(base_size = 12) +
theme(
plot.margin = margin(t = 8, r = 24, b = 8, l = 8),
axis.text.x = element_text(angle = 0, vjust = 1, hjust = 0.5),
plot.caption = ggtext::element_textbox_simple(margin = margin(t = 6)),
plot.caption.position = "plot"
) +
labs(caption = my_caption) +
coord_cartesian(clip = "off")
วิธีรายงานผล
รายงาน ความต่างเฉลี่ยที่สนใจ พร้อมค่า 95% CI และวิธี post-hoc test (เช่น Tukey) ใส่ขนาดเอฟเฟกต์ (เช่น partial η² หรือ ω²) เพื่อสื่อสาร